北京时间10月3日下午,诺贝尔基金会宣布将2018年的诺贝尔化学奖授予Frances H. Arnold教授、George P. Smith教授、以及Gregory P. Winter爵士,以表彰他们成功驾驭演化的力量,造福人类。奖项的一半授予美国科学家阿诺德(Frances H. Arnold),表彰她实现了酶的定向演化;另一半授予给美国科学家史密斯(George P. Smith)和英国科学家温特(Gregory P. Winter),表彰他们实现了多肽和抗体的噬菌体呈现技术。
工欲善其事,必先利其器。化学家也一样,他们在研究酶和蛋白质的时候,首先要把它们用三维的方式表达出来。
达索系统的BIOVIA就是化学家们常用的武器。BIOVIA的前身是Accelrys。
生物分析、药物发现、毒理学、疾病诊断和监测、食品安全等都极大的促进了蛋白质组及代谢组学的发展。但这也给研究者在获取和处理数据上提出了更高的要求。
Pipeline Pilot拥有众多的数据分析模拟,如蛋白质谱分析模块、序列分析模块、R统计模块。并能够自由组合成为工作流,提高研究者对蛋白质组学数据的分析、管理和共享能力。
1、蛋白质谱分析模块
Mass Specfor Proteomics模块提供了一系列对基于生物质谱的蛋白组数据处理工具,可以对来自质谱实验的数据进行读入、输出、显示、处理、分析、比较和发布。可以和Sequence Analysis和Gene Expression模块进行连用做其他实验数据的分析以及生物标记的识别和验证。
2、序列分析模块
实现对蛋白质和核酸的序列的注释和特征分析,序列搜索和比对,产生引物和扩增子,siRNA设计,发现限制性酶切位点等功能。同样,本模块可以通过Java和Perl建立新的组件来扩展系统功能,或通过与EMBOSS整合而实现功能的扩展。
3、 R统计模块
RStatistics模块能对数据进行深入细致的分析,创建内容丰富、清晰直观的分析图表,帮助您做出合理的决策。R统计模块包含的组件能够实现数据操作、聚类、学习,以及一些传统和探索性的数据分析方法,其统计引擎来自于在公共领域广泛应用的R统计学软件包。R统计学软件包中的数据统计和分析方法被无缝连接到PipelinePilot数据流程中,用户可以直接调用其组件而无需编写R脚本语言,所获得的输出结果可以直接用Pipeline Pilot工作流程中的其它组件进行分析。本模块中主要的学习方法有偏最小二乘法,神经网络和支持向量机等。R脚本语言编写的组件同样可以直接整合到Pipeline Pilot中,从而扩展R模块中的组件功能。
酶是自然界经过长期进化而产生的生物催化剂。它能在温和条件下高效专一地催化某些化学反应。正是它们的存在,确保了生物体内化学反应可以在常温,常压条件下进行。具有非常广阔的应用前景。而在近几年的国内外研究中,把分子模拟应用于酶领域成为了一个热门,研究手段也日益成熟。
应用模块:
Homology Modeling、Molecular Docking、Molecular Dynamics、QM/MM、ZDOCK/RDOCK、Protein Design
Arnold博士于1986年加入加州理工学院,曾担任客座副教授、助理教授、教授和主任。她的实验室侧重于通过定向进化开展蛋白质改造,应用在替代能源、化工和医学方面。
Arnold博士可真的是不简单,曾获得众多荣誉,包括进入美国发明家名人堂(2014)、美国发明家科学院院士(2014)、ENI可再生能源和非常规能源奖(2013)、美国国家技术与创新勋章(2011)以及美国国家工程院的Charles Stark Draper奖(2011)。她当选为美国三个国家研究院(科学院、医学院和工程院)以及美国艺术与科学院的院士。
用酶来催化有机反应并不新鲜,Frances Arnold教授堪称这一领域内的“女神”级人物。她课题组2016年一篇“酶法构建C-Si键”的Science工作,让不少人惊呼构建“硅基生命”终于成为可能。
就在今年,她的团队已经制造出一种细菌,可以制造出小型但充满能量的碳环,这些碳环是制造其他化学物质和材料的有用材料。这些“戒指”本来是特别难打造的,现在可以像啤酒一样“酿造”了。
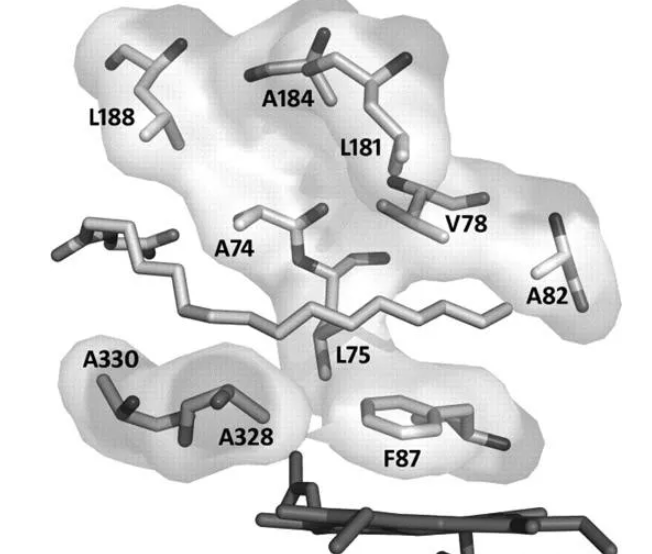
以下是一篇她的团队用BIOVIA作为工具,来表达分子式的文章。
Comparison of random mutagenesis and semi-rational designed libraries for improved cytochrome P450 BM3-catalyzed hydroxylation of small alkanes
随机诱变和半合理设计库比较改良细胞色素P450 BM3催化小烷烃羟基化
摘要:三种半合理方法,使用还原氨基酸组的组合位点饱和诱变(CSSM)和基于Corbit的两个库和针对多达10个活性位点残基的CRAM计算设计算法,用于改造细胞色素P450 BM3去甲基化二甲基醚和羟基丙烷和乙烷。与随机诱变文库和靶向相同残基的单个位点饱和库相比,这些小库(343-1028变体)在功能和最大活性部分方面都得到了丰富。尽管平均氨基酸取代水平高达2.6,5和7.5,但CSSM,Corbit和CRAM库至少有75%的库成员正确折叠。使用所有三种诱变方法鉴定丙烷 – 和乙烷 – 羟基化P450 BM3变体,具有少至两个氨基酸取代。使用CRAM算法设计的库试图减小结合口袋的大小,产生了更多数量的活性变体和变体,支持最大数量的催化转换。最活跃的变体E32在36%偶联时支持16 800个丙烷转换,其与使用随机和位点饱和诱变10-12轮定向进化后获得的变体的活性相媲美。该研究中没有变体实现了先前通过多轮诱变和筛选获得的丙烷羟化(包括93%偶联)的完全再特化。然而,这些半合理方法允许序列空间中的大跳跃到具有所需功能的变体。
烷烃羟基化反应
P450乙烷和丙烷羟基化反应在可加压的96孔反应器(Accelrys,USA)中进行。总体积为0.5ml的反应通常含有20-40μl过滤(2μm)粗细胞提取物或浓度为50-200nM的纯化蛋白,以及由20mM DL-异柠檬酸三钠盐组成的NADPH再生系统,400μMNADP+和0.5U /ml异柠檬酸脱氢酶在烷烃饱和的0.1M磷酸钾缓冲液中。用烷烃将反应器顶部空间加压至30psi,并将反应在4℃下搅拌至少20小时。加入15μl5MHCl淬灭反应,并用75μl1M磷酸钾缓冲液(pH8.0)中和。通过离心除去沉淀的细胞碎片和变性蛋白质后,将1-戊醇作为内标添加到反应混合物中,并通过气相色谱法用火焰离子化检测器测定醇产率。
Congratulations to Frances, Georgeand Gregory!
由 3DEXPERIENCE® 平台支持,达索系统的BIOVIA™可提供全球协作产品生命周期体验,以此改变科学创新。
行业领先的 BIOVIA 产品组合致力于集成整个研究、开发、QA/QC 和制造过程中的科学、实验流程和信息要求。功能包括科学数据管理;小分子、生物制剂和材料建模和仿真;化学和生物信息学;系统生物学和一体化治疗学;协作网络研究;科学流水线技术;企业实验室管理;法规和质量管理;流程知识和协作以及化学品库存管理。